DNA作为天然的信息载体,凭借超高存储密度、抗电磁干扰、长期稳定性等优势,已成为冷数据(低频访问、长期归档)存储的重要候选方案。但现有DNA存储技术多依赖序列合成的顺序存储模式,无法实现热数据(高频访问、动态修改)所需的快速随机访问和灵活编辑功能,严重限制了其应用场景。
为了解决这些问题,近日麻豆视频 晁洁教授团队提出一种基于DNA折纸纳米结构的链式数据存储(DONLDS)系统。该系统创新性地将链表数据结构与DNA纳米技术相结合,成功实现英文、数字、中文等多类型数据的高效存储、动态插入与删除,为下一代信息存储技术开辟了新路径。相关工作以“Linked data storage using DNA origami nanostructures”为题,发表在《Nature Communications》期刊。
科研团队借鉴计算机链表存储架构,设计了三种具有特定形状的DNA折纸纳米结构(DONs)作为存储节点,其中三角形(TDON)、矩形(RDON)和十字形(CDON),分别承担不同类型数据的存储任务。每个节点集成了数据域和指针域:数据域通过链霉亲和素与生物素修饰的数据链结合,以二进制形式编码信息;指针域则由边缘特定序列的DNA链构成,通过与可分离的“指令链”互补配对,实现节点间的精准连接与断开。这种设计巧妙借鉴了计算机科学中的链式数据存储思想,无需连续的存储单元,可根据数据大小动态分配空间,避免了传统存储中数据修改时的大规模迁移。
DONLDS系统展现出强大的多模态数据存储能力,首先研究团队基于TDON节点成功编码存储了26个英文字母,实现“HELLO WORLD”字符串的精准链接。定量分析结果显示,SA能够精准锚定于预设的数据域位置,各TDON的信息存储准确率分布于91.17±0.86% 至 95.64±2.05%之间。这一高精度且波动范围较小的实验结果,充分证实了基于TDON的信息编码方案具备优异的可靠性与操作稳定性,为其在高密度数据存储或分子编码领域的实际应用奠定了实验基础。
更值得关注的是,该存储系统具备灵活的动态数据编辑能力。研究团队通过调控指令链的连接与断开,成功演示了数据的插入与删除操作:将存储“DNA helix 1953”的链表结构,通过删除“helix”和“1953”节点、插入“storage”和“1988”节点,精准转化为“DNA storage 1988”,整个过程无需遍历全部数据结构,操作效率大幅提升。经24个样本的交叉验证,数据可完全恢复,充分证明了其编辑准确性。这些结果进一步证实了基于DNA纳米技术的数据处理系统在信息存储与操作领域的应用潜力。
该工作得到了国家重点研发计划、国家自然科学基金、江苏省自然科学基金前沿技术项目及新基石科学基金会的联合支持。有机电子与信息显示国家重点实验室的晁洁教授为论文的通讯作者,博士生张晨昊为论文的第一作者。谢茉副教授,汪联辉教授,樊春海院士,为该研究工作做出了重要贡献。
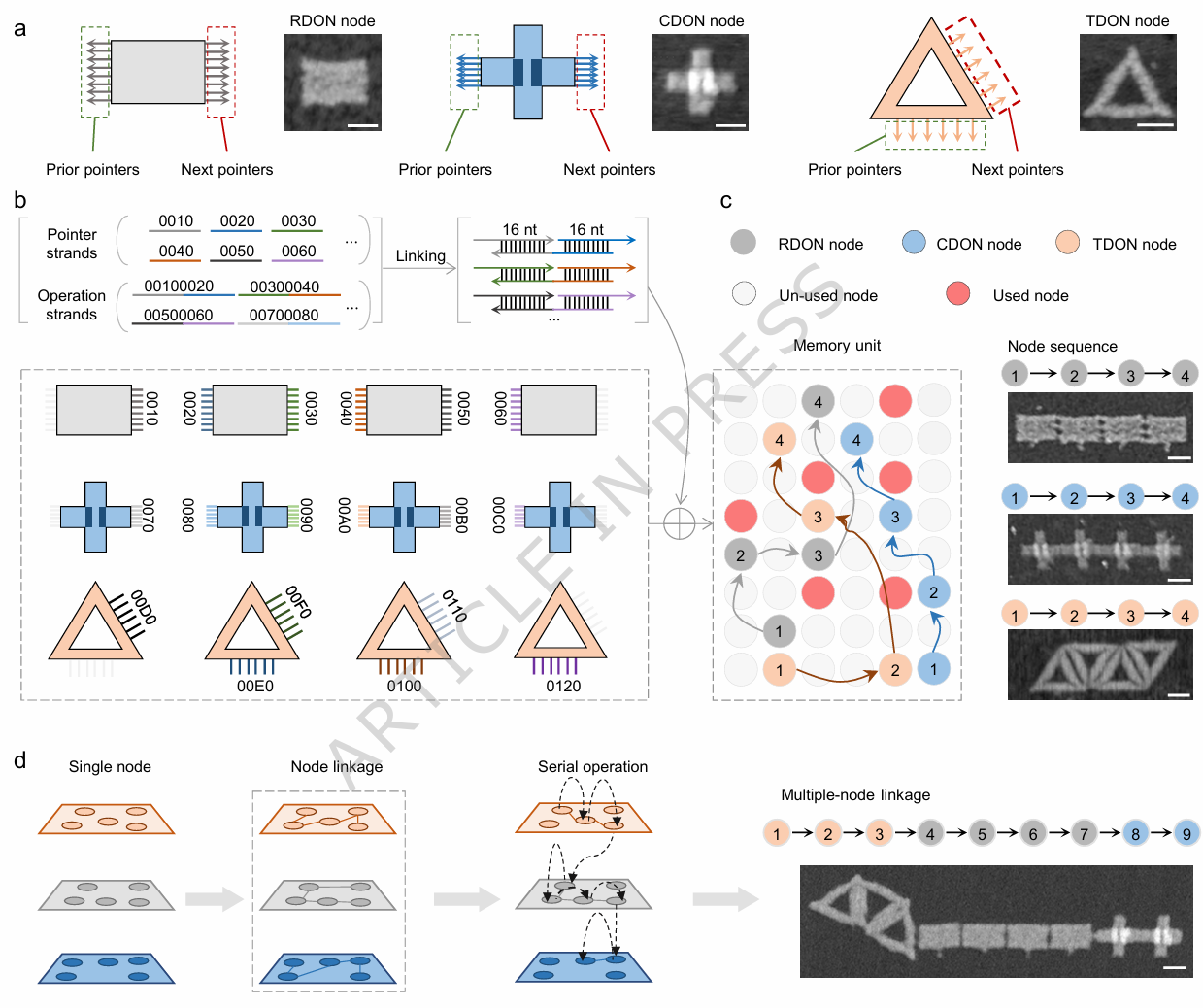
基于DNA折纸纳米结构的链式数据存储(DONLDS)系统概述图
原文链接://www.nature.com/articles/s41467-025-66274-x
(撰稿:谢茉 编辑:陈宁娜 审核:凌海峰)

